Usługa jest przeznaczona dla biologów i bioinformatyków.
Usługa BioKepler składa się z kilku elementów: dostarczane jest całe wirtualne środowisko pracy dostępne poprzez zdalny desktop: maszyna wirtualna uruchomiona na zasobach chmurowych, a na niej zainstalowany zestaw popularnych narzędzi i aplikacji bioinformatycznych oraz oprogramowanie BioKepler. BioKepler jest modułem platformy zarządzania naukowymi scenariuszami obliczeniowymi - Keplera, który służy do uruchamiania zestawu narzędzi bioinformatycznych w rozproszonych środowiskach obliczeniowych takich jak PLGrid. Użytkownik może tworzyć całe scenariusze obliczeniowe (ang. workflows lub pipelines), składające się z wielu kroków, a poszczególne komponenty mogą być uruchamiane na różnych zasobach obliczeniowych.
Przykładowe komponenty BioKeplera (tzw. aktorzy): 2bwt-builder, DynamicTrim.pl, RNA_parse.pl SOAPdenovo31mer, blast_rRNA.pl, blastall, bowtie, bowtie-build, bowtie2-build, breakdancer-max, bwa_align bwa_index, cd-hit cd-hit-454, cd-hit-est, clustalw, clustalx, cuffdiff, cytoscape, faa_stat.pl, fastq2fasta.py, fraggene_scan.pl, fraggene_scan_parse.pl, hmm_rRNA.pl, hmmsearch.pl, hmmsearch_parse.pl, kegg.pl, kegg_parse.pl, metagene.pl, mother, muscle orf_2_tbl.pl, orf_finder, qc_filter.pl, qc_filter_fastq.pl, qiime rasmol, rpsblast.pl, rpsblast_parse.pl, samtools, soap, ssake, tRNAscan-SE.pl, tophat, uchime, velvetg, velveth
Środowisko Kepler, w którym scenariusze są tworzone graficznie poprzez łączenie aktorów (elementarnych cegiełek wykonujących określone zadanie) przy użyciu kabli (które transportują dane), ma wbudowaną bibliotekę ponad 400 aktorów (np. 'wyrażenie matematyczne', 'czytnik danych tabelarycznych', 'kod Java', 'skrypt Python', 'narysuj wykres'). Gotowe scenariusze mogą być uruchamiany w trybie graficznym bądź z linii komend.
Aby skorzystać z usługi BioKepler, należy mieć aktywne konto w Infrastrukturze PLGrid oraz aktywną afiliację.
Do usługi BioKepler może uzyskać dostęp każdy użytkownik PLGrid, który jest użytkownikiem usługi Molecular Biology Data Analysis Toolkit . W celu uzyskania dostępu do tej usługi należy po zalogowaniu się w Portalu PLGrid w lewym menu wybrać zakładkę Usługi. Po przejściu do tej zakładki należy kliknąć zielony przycisk Zarządzaj usługami, znajdujący się w prawym górnym rogu. Spowoduje to przejście do Katalogu Aplikacji i Usług (KAiU), gdzie należy wyszukać usługę Molecular Biology Data Analysis Toolkit, a następnie o nią aplikować.
Przyznanie usługi następuje automatycznie. Gdy usługa zostanie przyznana, pojawi się na liście usług w portalu PLGrid ze statusem „active” Status usługi będzie również widoczny w KAiU jako "Status użytkownika: Usługa aktywna — usługa dostępna dla użytkownika".
Poniżej przedstawione są następujące kroki:
BioKepler62.3.168.25plgusernameGNOMEUwaga: można stworzyć sobie poprzez X2Go kilka sesji, na raz może być aktywna tylko jedna z nich.
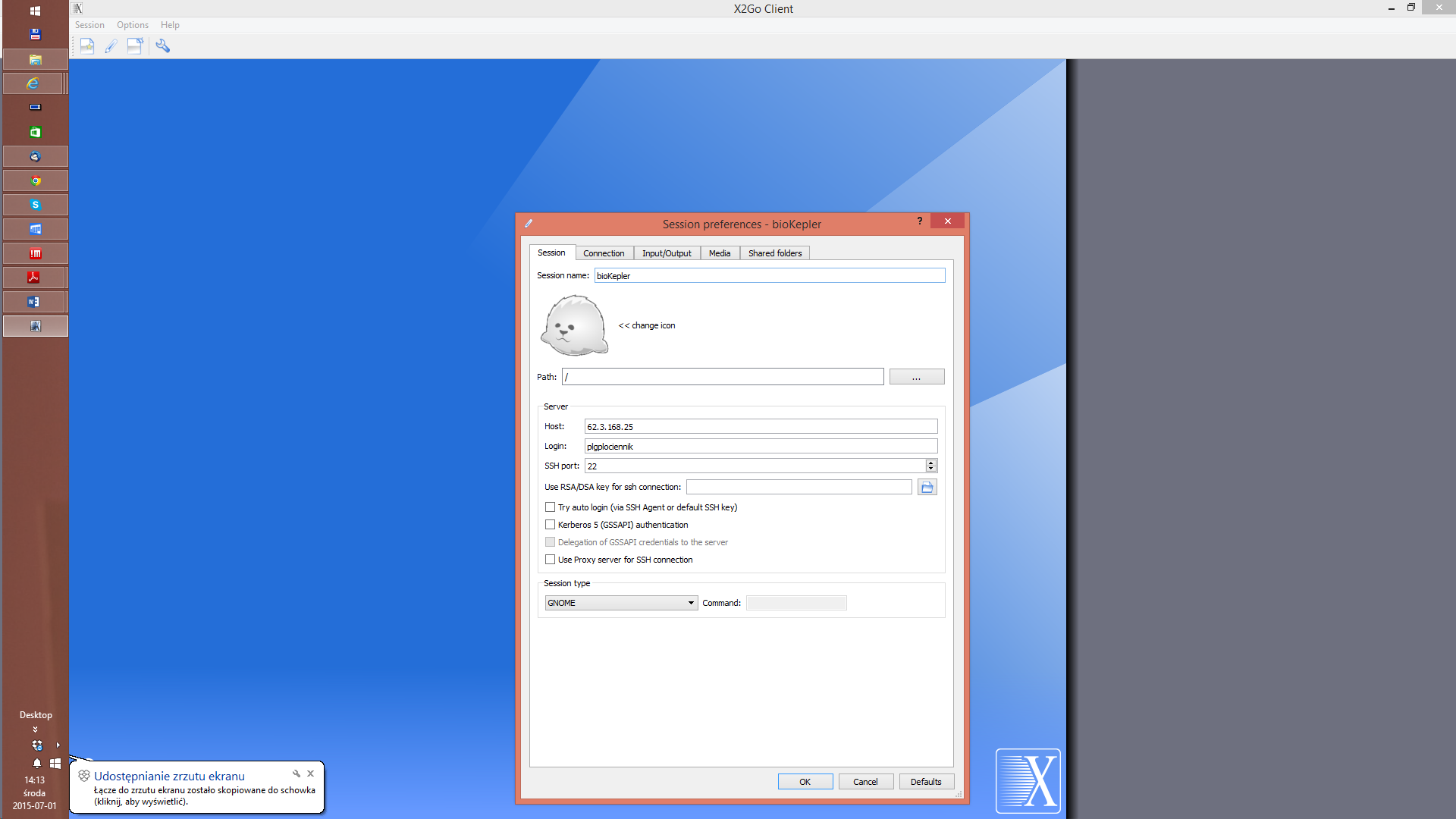
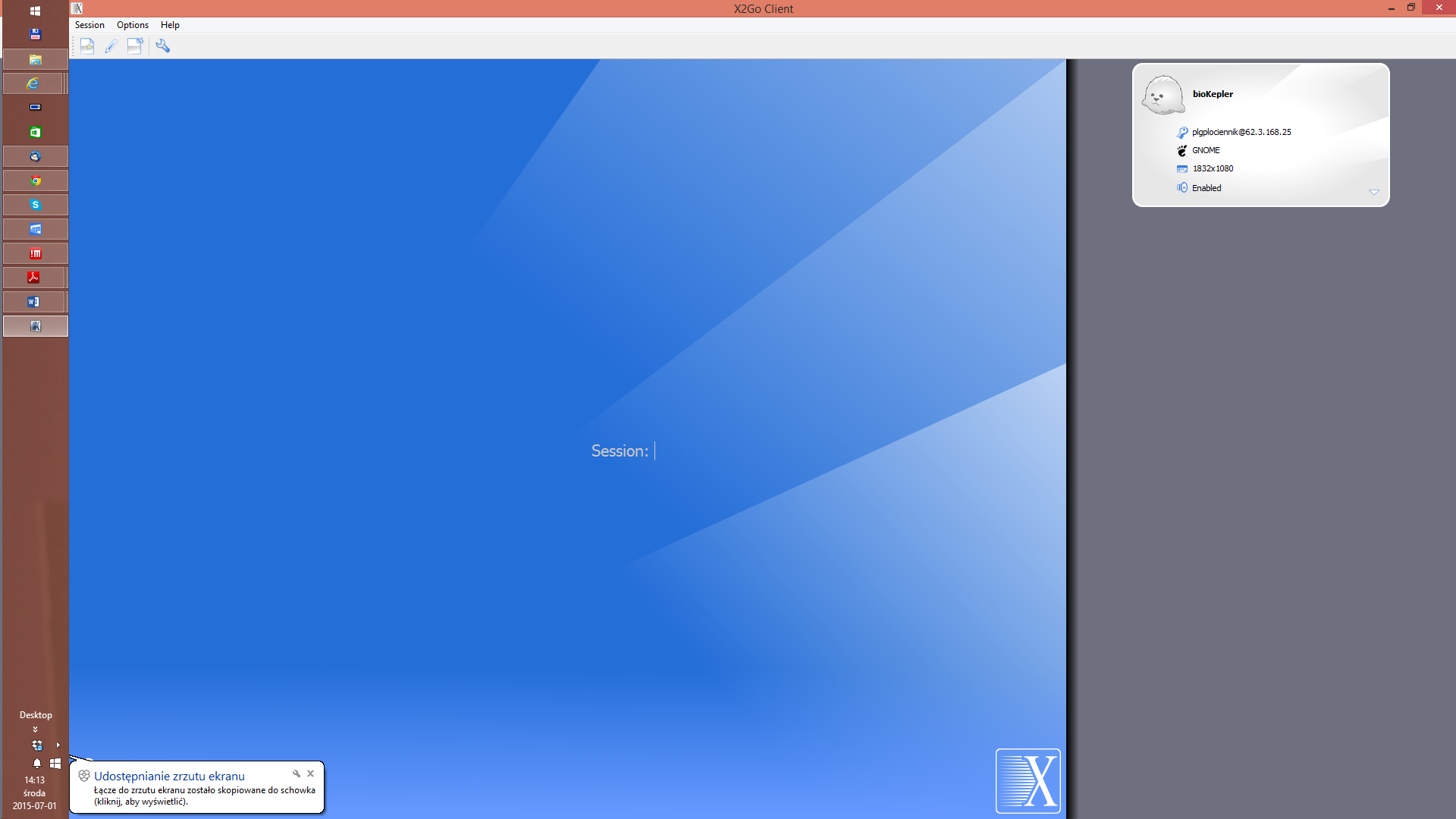
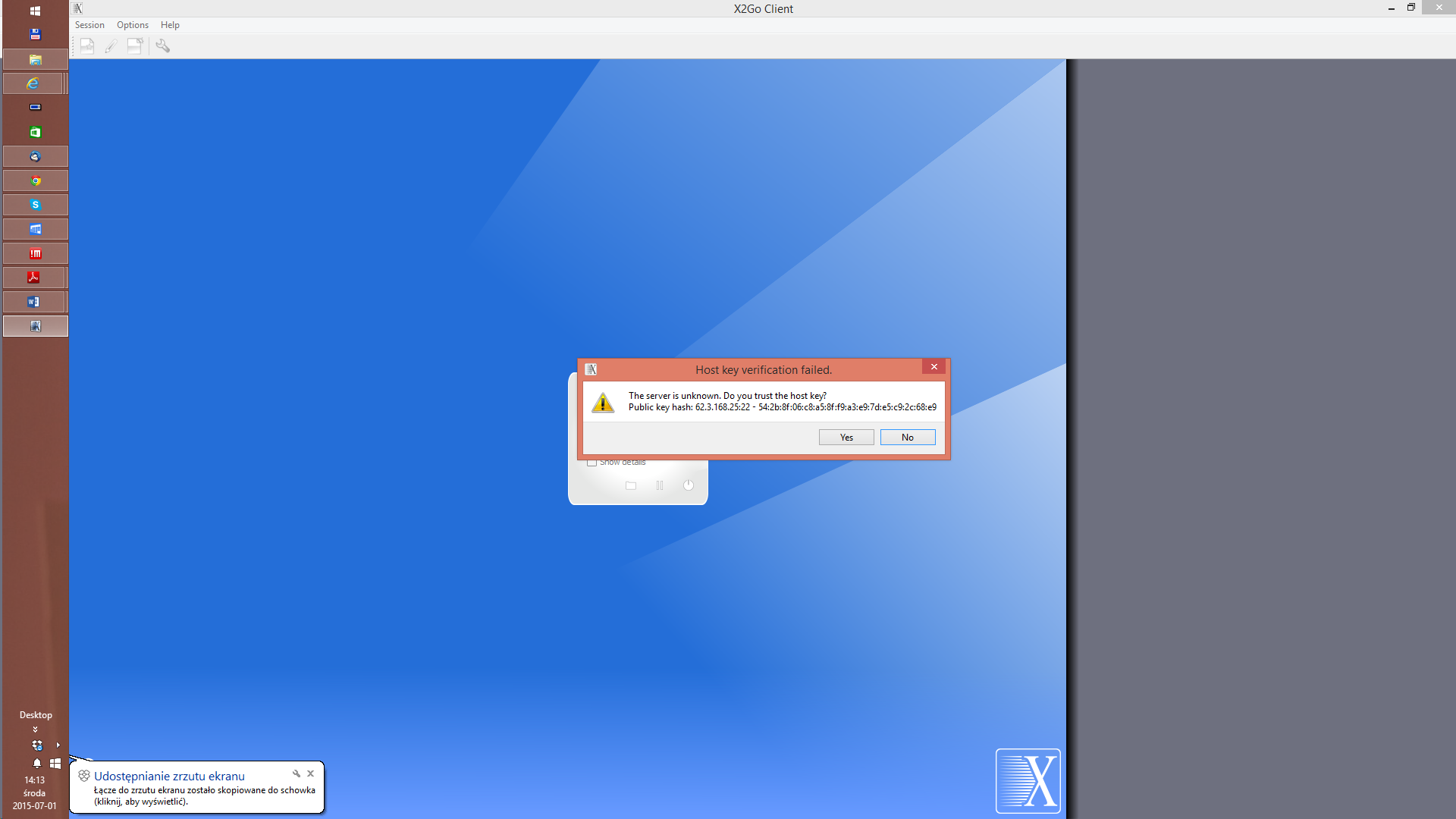
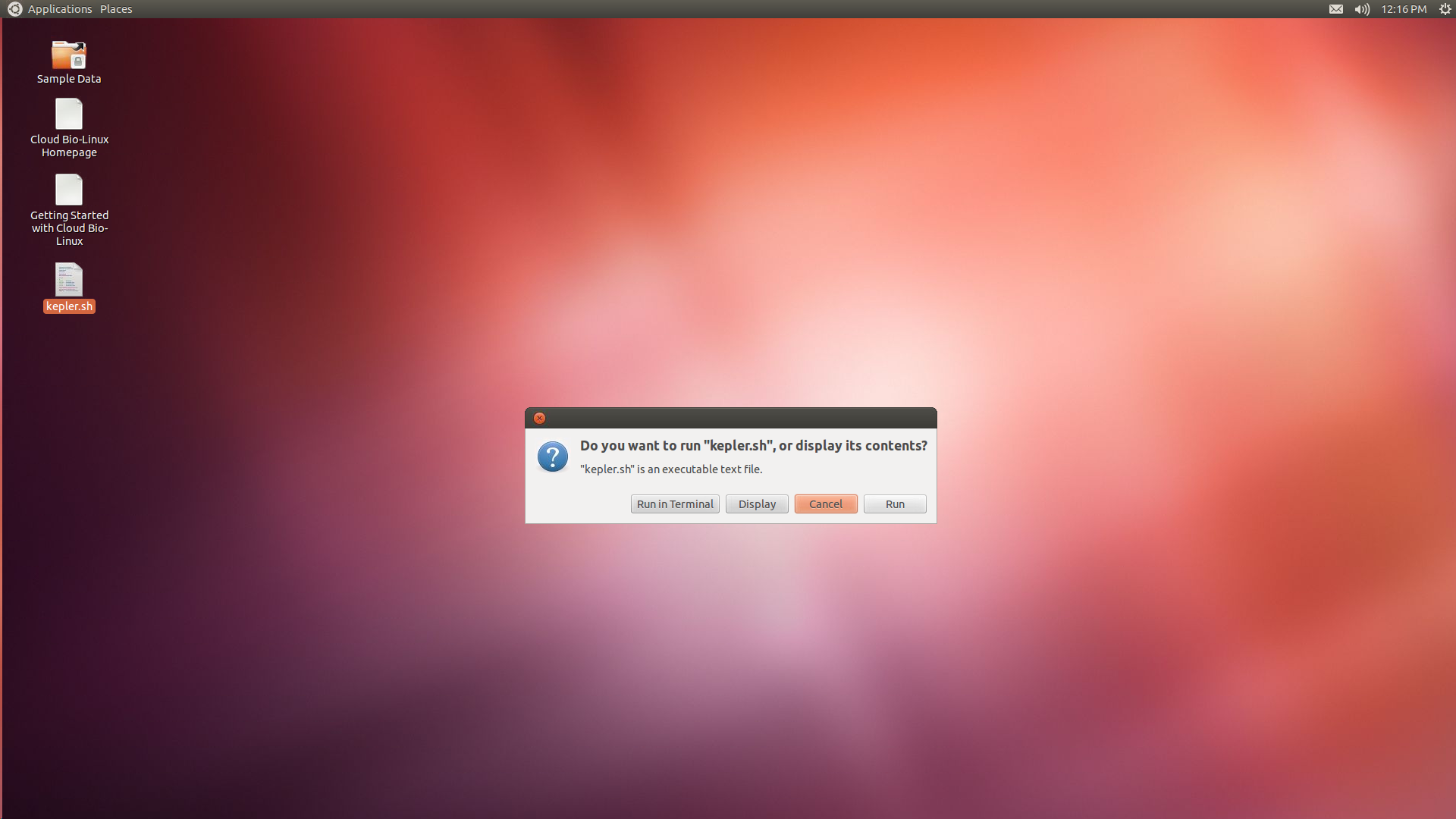
Po połączeniu ze zdalnym środowiskiem pracy, powinien pojawić się zdalny pulpit, na której między innymi znajduje się ikona do narzędzia BioKepler: kepler.sh

"Run in terminal" da możliwość obserwowania komunikatów w konsoli.
Pierwsze uruchomienie BioKeplera będzie trwało dłużej ze względu na automatyczne tworzenie katalogów i kopiowaniu plików do katalogu domowego..
Po uruchomieniu pojawi się ekran powitalny aplikacji, który należy zamknąć(można zaznaczyć na dole okna, żeby nie pojawiał się przy następnych uruchomieniach).
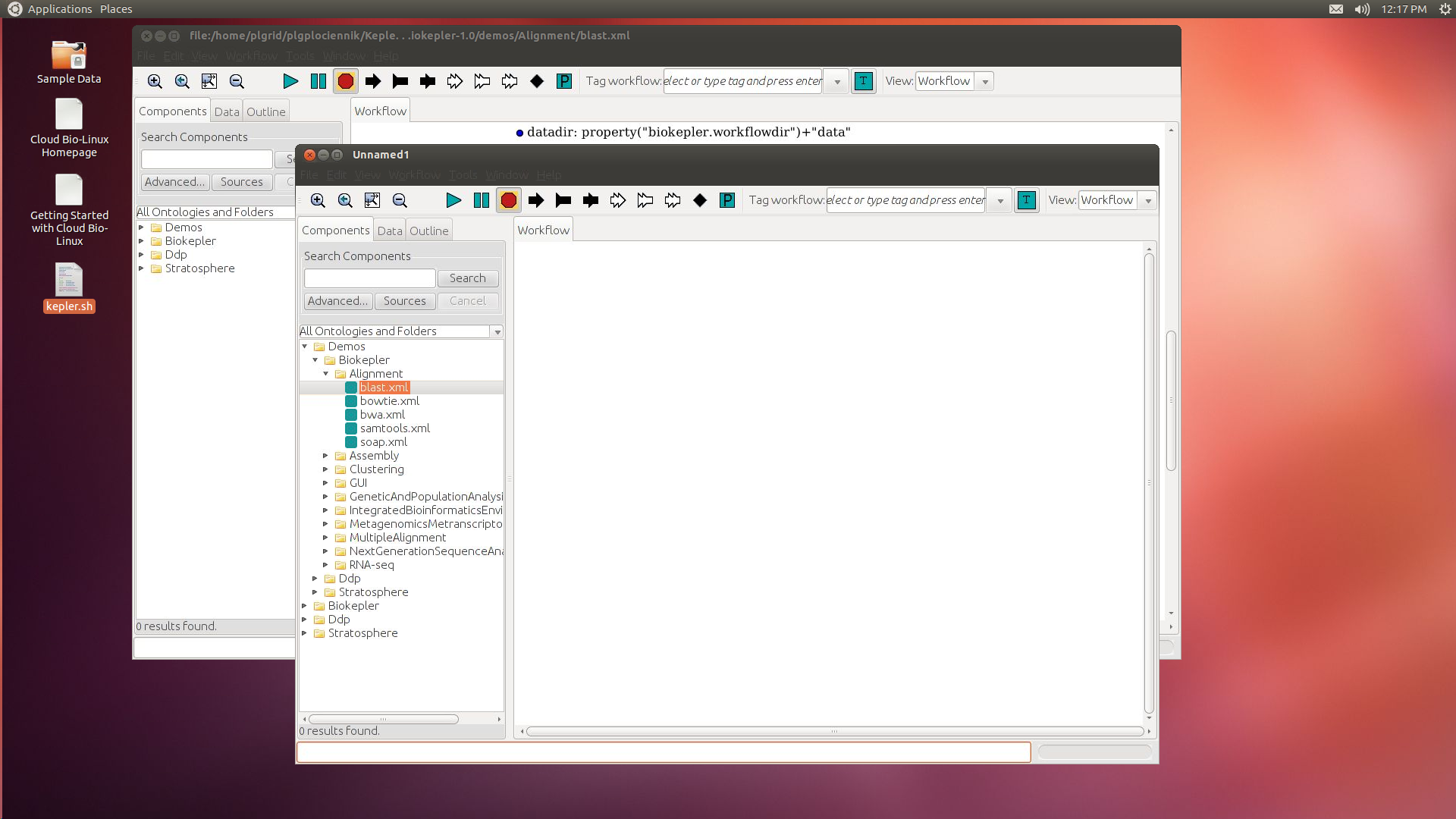
Z zakładki po lewej stronie przestrzeni roboczej (dashboard) można wybrać gotowe scenariusze lub utworzyć samemu nowy scenariusz. Dla przykładu wpisując "blast" lub wybierając "Demos BioKepler Alignment Blast.xml"
i klikając podwójnie na ikonę można otworzyć przykładowy workflow
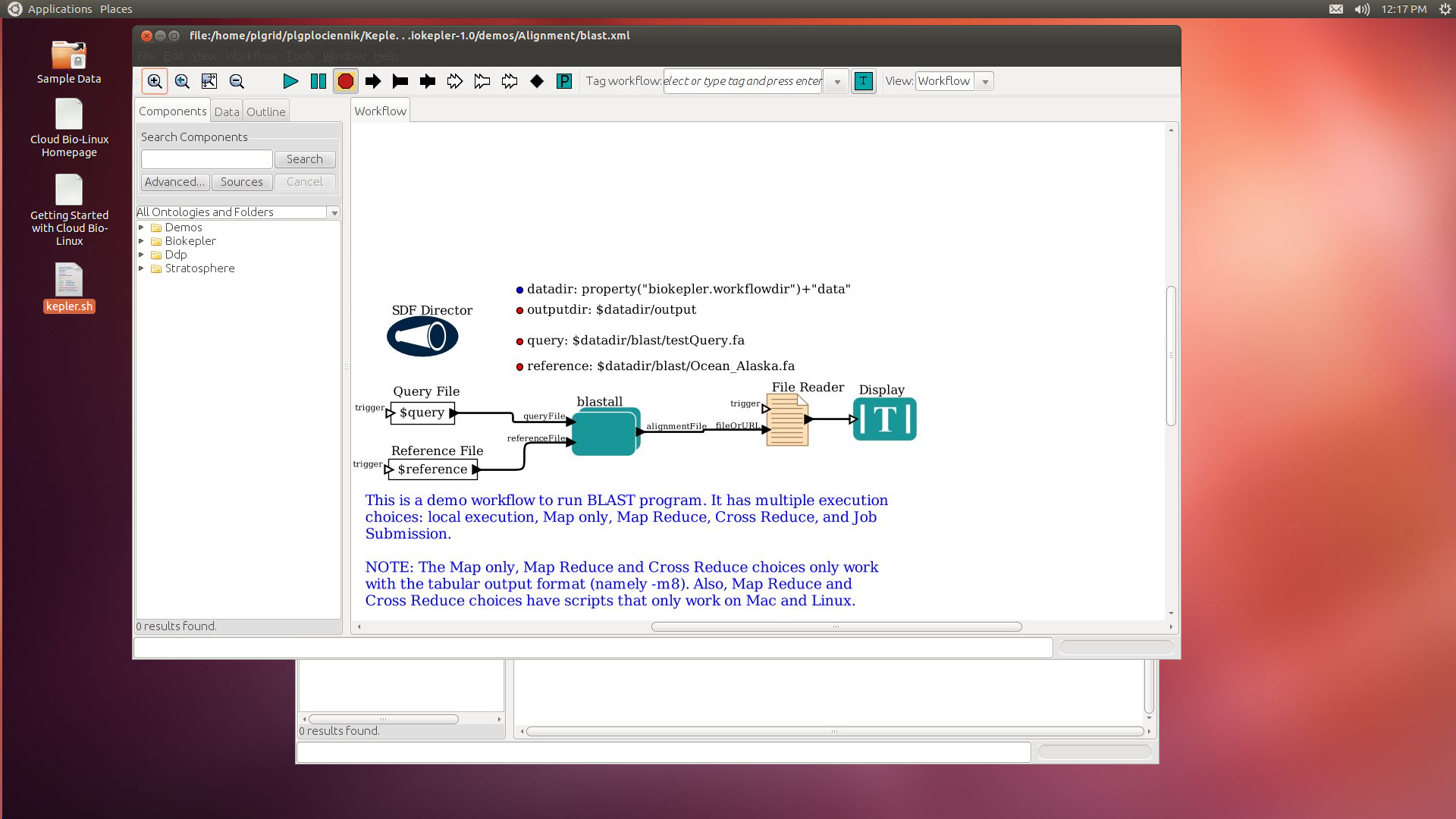
Ponieważ workflow jest prekonfigurowany i są dostępne przykładowe dane wejściowe, można od razu przejść do uruchomienia scenariusza.
Można to osiągnąć poprzez naciśnięcie ikony "press"
Wynik uruchomienia tego scenariusza powinien się pojawić w osobnym oknie (blast.Display)
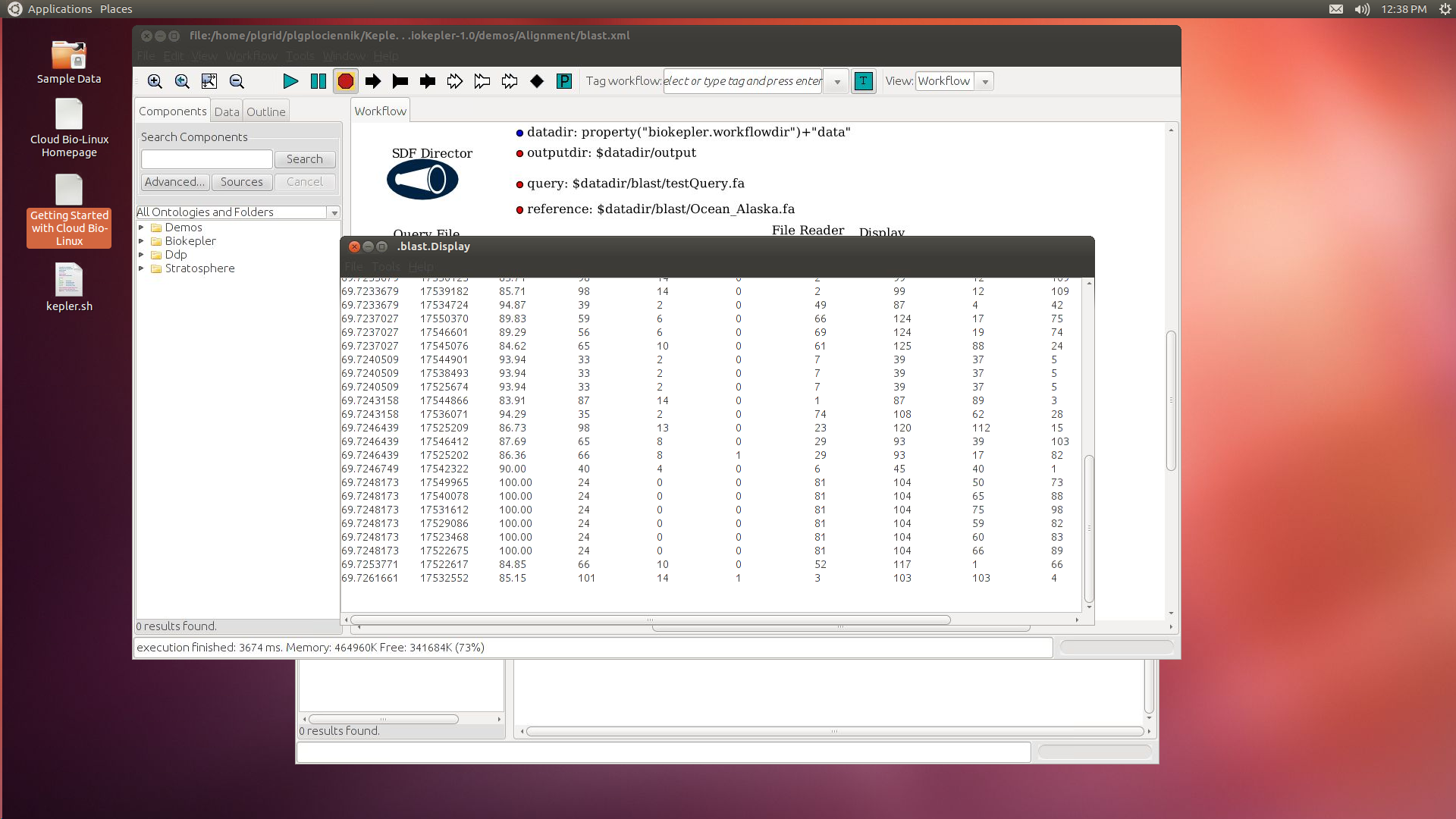
Analogicznie do przykładu przedstawionego w sekcji pierwsze kroki, można uruchomić kilkanaście innych gotowych scenariuszy.
Poniżej przykładowe wyniki uruchomień przykładowych scenariuszy:
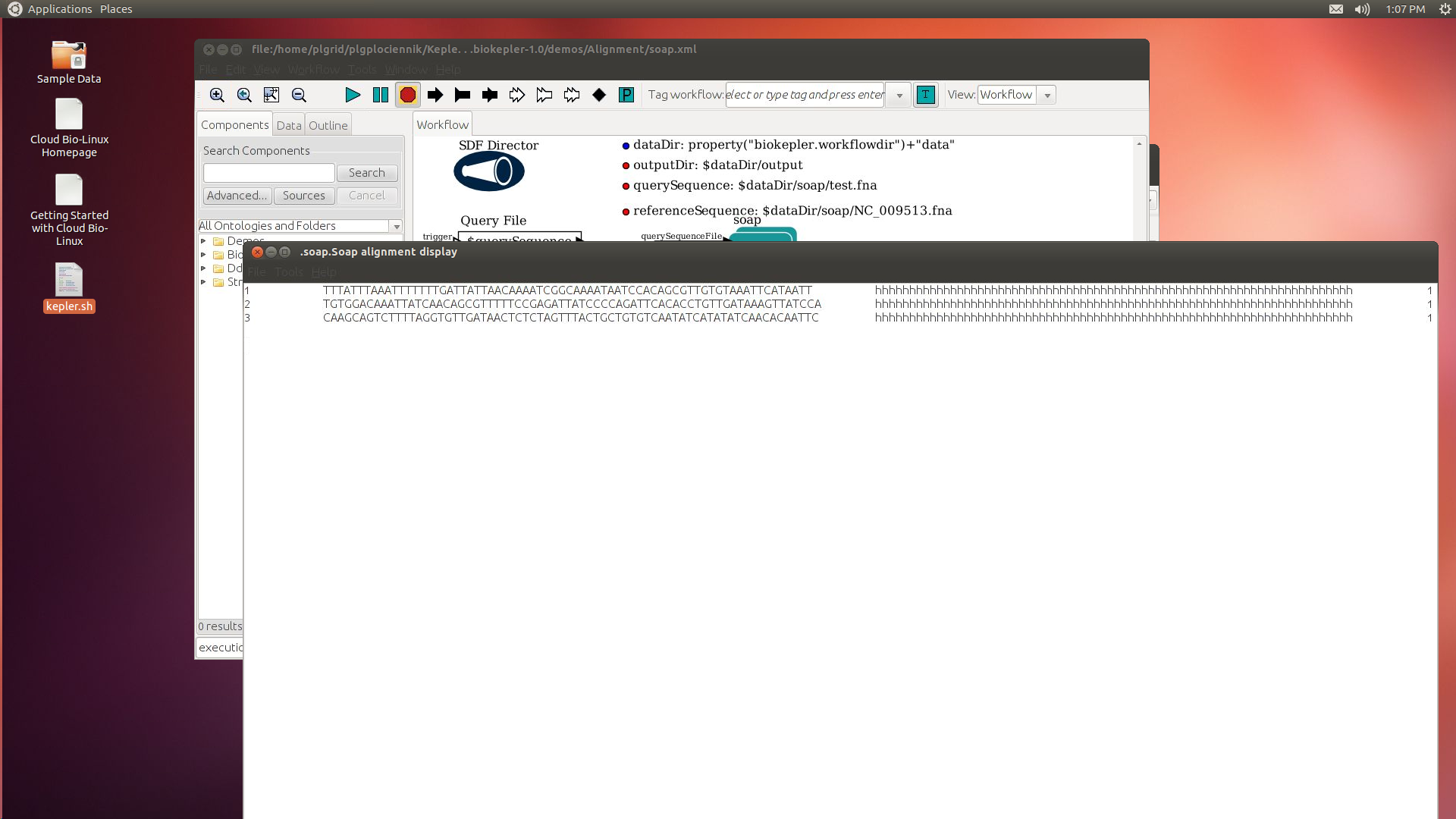
Soap
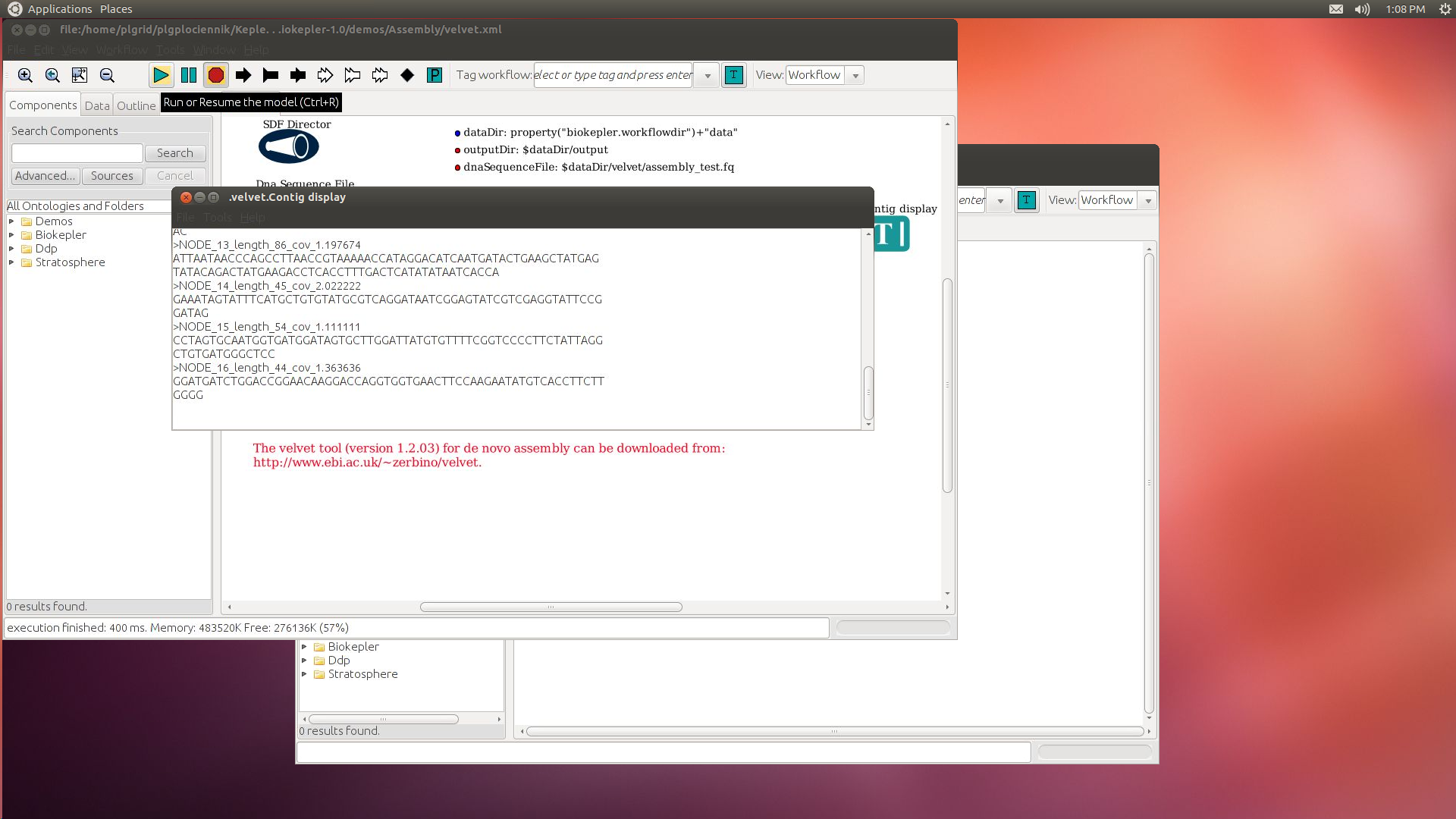
Assembly/velvet
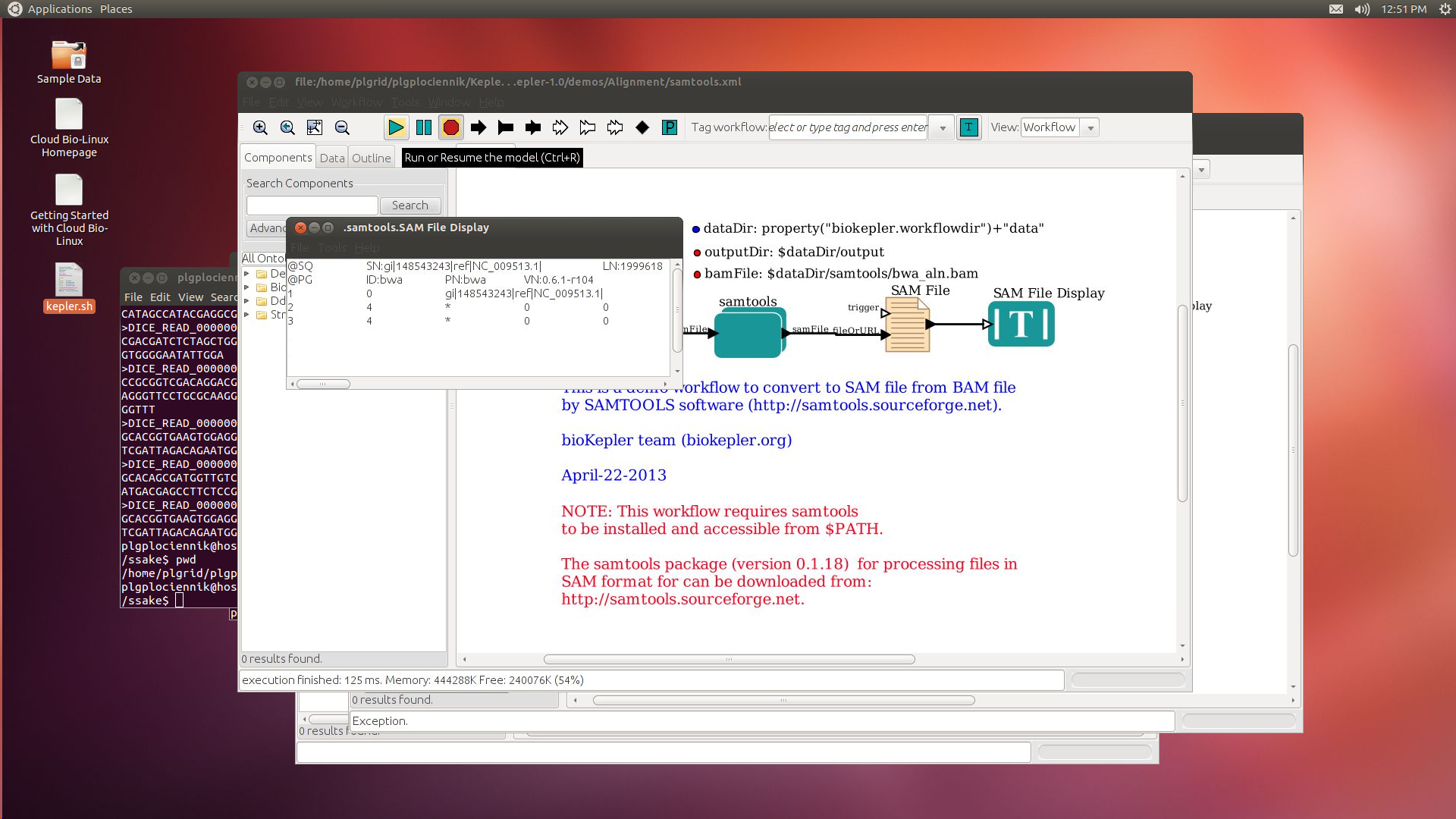
SAMTOOLS
Zestaw kilkuset najczęściej używanych programów domenowych jest dostępnych na ścieżce uruchomieniowej, może być również wykorzystany poprzez BioKeplera.
Część narzędzi bioinformatycznych jest również dostępnych z poziomu menu (na przykład: Dendroscope, cn3d, sequin, splitstree, tetra, trev, squint, etc)
W sprawie konstruowania innych scenariuszy/pomocy przy konfigurowaniu obecnych prosimy kontaktować się przez helpdesk PLGrid.
Szczegółowe informacje o użytkowaniu infrastruktury PLGrid znajdują się w Podręczniku Użytkownika.
Szczegółowa dokumentacja programu BioKepler znajduje się na stronie: http://www.biokepler.org/userguide
Szczegółowa dokumentacja platformy Kepler znajduje się w pliku: dokumentacja.pdf
Informacje o usługach dziedzinowych Biologia dostępne są na stronie: http://biologia.plgrid.pl/
Uzyskanie informacji/helpdesk PLGrid: dokumentacji o pomocy